Qualitätsbewertung von MALDI-Imaging-Daten
| Arbeitsgruppe: | AG Technomathematik |
| Leitung: | Prof. Dr. Dr. h.c. Peter Maaß ((0421) 218-63801, E-Mail: pmaass@math.uni-bremen.de ) |
| Bearbeitung: |
Delf Lachmund
Dr. Janina Oetjen Dr. Tobias Boskamp |
| Projektpartner: | |
| Laufzeit: | seit 01.04.2015 |

Massenverschiebungsprofil einer MALDI-Imaging-Messung. Punkte markieren die Abweichungen gegenüber dem theoretischen Peptidmodell für lokale Maxima des Mittelwertspektrums. Die rote Mittellinie gibt die geschätzte Massenverschiebung an, die schwarz gestrichelten Linien den erwarteten Referenzkorridor.
Mit der Methode der MALDI-Imaging-Massenspektrometrie werden räumlich aufgelöste Spektraldaten akquiriert, die bei einer Ortsauflösung von wenigen Mikrometern mehrere hunderttausend Spektren umfassen können. Jedes Spektrum besteht wiederum aus bis zu 40.000 Datenpunkten, sodass bereits eine einzige Messung eine große Menge an Daten liefert.
Neben den eigentlich relevanten Informationen über die jeweiligen Konzentrationen der in der untersuchten Probe vorliegenden Substanzen weist ein MALDI-Imaging-Datensatz eine Vielzahl von Signalstörungen und -überlagerungen auf, die die Auswertung der Daten und deren Vergleichbarkeit stark einschränken. Aufgrund der Komplexität der Daten und der unterschiedlichen Störeinflüsse, die in hohem Maße von der Probenpräparation und der Durchführung der MALDI-Messung abhängen, existiert bislang keine praktikable, objektive und breit anerkannte Methode, die Qualität eines MALDI-Imaging-Datensatzes zu bewerten. Eine solche Methode ist jedoch für eine Optimierung der Datenakquisition und die Festlegung einer Standard Operating Procedure (SOP) unerlässlich.
Aufbauend auf Arbeiten der früheren ZeTeM-Mitarbeiter Theodore Alexandrov und Andrew Palmer wurde in der ZeTeM-Gruppe Bioinformatik eine Methodik zur Qualitätsbewertung von MALDI-Imaging-Daten und zur systematischen Entwicklung von anwendungsspezifischen SOPs zur Datenakquisition entwickelt. Zentrale Elemente dieser Methodik sind a) die Festlegung eines teilfaktoriellen Experimentplans (fractional factorial design of experiments) zur Durchführung einer Folge von Vergleichsmessungen, sowie b) die Nutzung eines linearen ANOVA-Modells (Analysis of Variance) zur Bewertung der Einflüsse der verschiedenen Akquisitionsparameter auf die Ergebnisqualität. Für die Qualitätsbewertung werden einerseits bildliche Strukturmaße verwendet, wie das von Alexandrov und Palmer entwickelte Measure of Chaos, sowie andererseits modellbasierte Maße, die die Übereinstimmung charakteristischer Datenmerkmale mit einem theoretischen Modell bestimmen.
Zur zweiten Kategorie gehört eine jüngst entwickelte Methode, Massenverschiebungen in MALDI-Imaging-Daten zu visualisieren und zu quantifizieren, die insbesondere für proteomische Analysen an histopathologischen Gewebeproben geeignet ist. Hierfür werden eine große Zahl von unspezifischen spektralen Merkmalen aus den Daten extrahiert und deren Massenzahlen mit einem einfachen theoretischen Modell verglichen. Aus der statistischen Verteilung der Abweichungen lässt sich das Ausmaß der Massenverschiebungen quantitativ ableiten sowie eine Korrektur der Daten vornehmen.
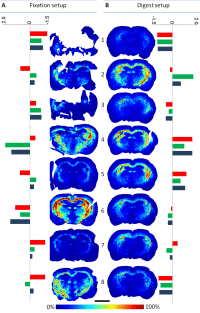
Bild rechts: Ionenbilder zum jeweils gleichen Peptid des Myelin-Basisproteins, extrahiert aus je acht Messungen aus zwei experimentellen Versuchsplänen (Fixation und Digest Setup). Die farbigen Balken zeigen die Werte von drei verschiedenen Qualitätsmaßen als Abweichungen vom jeweiligen Mittelwert.