BMBF-MaDiPath: Massenspektrometrisches Profiling/Grading für onkologische Routineanwendungen der digitalen Pathologie
| Arbeitsgruppe: | AG Technomathematik |
| Leitung: | Prof. Dr. Dr. h.c. Peter Maaß ((0421) 218-63801, E-Mail: pmaass@math.uni-bremen.de ) |
| Bearbeitung: |
Dr. Jens Behrmann
Dr. Tobias Boskamp Dr. Yovany Cordero Hernandez Dr. Christian Etmann Dr. Lena Hauberg-Lotte Delf Lachmund Dr. Janina Oetjen Sabine Eifeld (E-Mail: eifeld@math.uni-bremen.de ) |
| Projektförderung: | BMBF, KMU-innovativ: Medizintechnik |
| Projektpartner: |
Proteopath GmbH SCiLS, Zweigniederlassung der Bruker Daltonik GmbH |
| Laufzeit: | 01.10.2015 - 30.09.2018 |
Das angewandte Forschungsprojekt MaDiPath untersucht die Erforschung und Etablierung massenspektrometrischer Methoden, hier MALDI Imaging, für die digitale Pathologie. Ziel des Projekts ist es, Methoden zu entwickeln, die eine objektive, reproduzierbare und automatisierte Grundlage zur pathologischen Tumordiagnostik und der darauf aufbauenden personalisierten Verlaufs- und Therapieplanung ermöglichen. Es wird ein Ansatz verwendet, der auf proteomisch relevanten und a priori identifizierten Mustern von Mamma- und Ovarialkarzinomen aufbaut. Diese Tumoren haben eine große gesellschaftliche und medizinische Relevanz: Brustkrebs ist in Deutschland die häufigste Krebsart bei Frauen. Die Neuerkrankungen bei Eierstockkrebs sind zwar rückläufig, allerdings ist die Rezidivrate hoch und die Heilungschancen für betroffene Frauen gering.
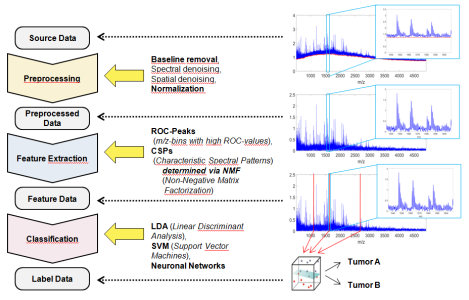
Ziel des Teilvorhabens des ZeTeM ist die Entwicklung eines NMF-Verfahrens zur Bestimmung charakteristischer spektraler Peptidmuster zur Nutzung in Klassifikations- und Regressionsmodellen für Tumoren aus MALDI-Imaging-Daten. In diesem Projekt wird ein wissensbasierter Ansatz verfolgt, bei dem spektrale Muster für bekannte Tumormarker modelliert und als Ausgangspunkt für eine datenspezifische Merkmalsextraktion verwendet werden (Targeted Feature Extraction).
Auf der Seite der mathematischen Forschung ist die Blind Source Separation mit Sparsity von besonderer Bedeutung: Blind Source Separation ist eine allgemeine Methode, um Daten voneinander zu trennen, die aus einer Überlagerung verschiedener Strukturen entstanden sind. Dieses Verfahren wird mit Sparsity-Ansätzen kombiniert, d.h. unter allen möglichen Überlagerungen, die die Daten hinreichend genau approximieren, werden diejenigen Lösungen bevorzugt, die mit den wenigsten Koeffizienten auskommen. Dadurch können Strukturen mit kleinen Amplituden, die eigentlich unterhalb des Rauschlevels der gemessenen Daten liegen, trotzdem zuverlässig getrennt werden. Übertragen auf die bildgebende Massenspektrometrie können dadurch die sehr komplexen Einzelspektren als Überlagerung einiger weniger spektraler Basismuster dargestellt werden. Zu diesem Ansatz, der über NMF-Verfahren mit Sparsity oder LASSO-Algorithmen im Projekt numerisch realisiert werden soll, gibt es inzwischen erste Publikationen, die deren Potential für die Pathologie belegen.
Publikationen
- J. Leuschner, M. Schmidt, P. Fernsel, D. Lachmund, T. Boskamp, P. Maaß.
Supervised Non-negative Matrix Factorization Methods for MALDI Imaging Applications.
Bioinformatics, bty909 , 2018. - Y. Cordero Hernandez, T. Boskamp, R. Casadonte, L. Hauberg-Lotte, J. Oetjen, D. Lachmund, A. Peter, D. Trede, K. Kriegsmann, M. Kriegsmann, J. Kriegsmann, P. Maaß.
Targeted Feature Extraction in MALDI Mass Spectrometry Imaging to Discriminate Proteomic Profiles of Breast and Ovarian Cancer.
Proteomics - Clinical Applications, 1700168 , Wiley, 2018. - J. Behrmann, C. Etmann, T. Boskamp, R. Casadonte, J. Kriegsmann, P. Maaß.
Deep Learning for Tumor Classification in Imaging Mass Spectrometry.
Bioinformatics, 34(7):1215-1223, Oxford University Press, 2018. - T. Boskamp, D. Lachmund, J. Oetjen, Y. Hernandez-Cordero, D. Trede, P. Maaß, R. Casadonte, J. Kriegsmann, A. Warth, H. Dienemann, W. Weichert, M. Kriegsmann.
A new classification method for MALDI imaging mass spectrometry data acquired on formalin-fixed paraffin-embedded tissue samples.
BBA - Proteins and Proteomics, , 2016.

