Entdeckung von Biomarkern in MALDI-TOF Massenspektrometrie mittels diskreter Wavelettransformation
| Arbeitsgruppe: | AG Technomathematik |
| Leitung: | Dr. Fedor Alexandrov |
| Bearbeitung: | Dr. Fedor Alexandrov |
| Projektpartner: |
Prof. Dr. Herbert Thiele, Bruker Daltonik GmbH, Bremen Prof. Dr. Andre M. Deelder, Leiden University Medical Center, the Netherlands |
| Laufzeit: | 15.02.2007 - 31.03.2010 |
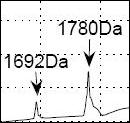
In Zusammenarbeit mit Bruker Daltonik GmbH und dem Medical Center der Universität von Leiden (Niederlande) soll ein Verfahren zur Entdeckung von Biomarkern in Massenspektrometrie-Proteinprofilen entwickelt werden. Dazu werden Verfahren diskreter Wavelet-Transformation und Support Vector Machines verwendet. Das Datenmaterial von Darmkrebs- und Bilharziose-Erkrankungen wurde im Medizinischen Center der Universität Leiden erfasst und mit MALDI-TOF-Massenspektrometern der Firma Bruker Daltonik gemessen.
Die Verwendung von Massenspektrometrie in der Krebsdiagnostik für die Suche nach Biomarkern - üblicherweise Peptide, die mit dem Infektionslevel korrelieren - wurde von E. F. Petricoin im Jahr 2002 im Journal „Lancet“ vorgestellt, erwies sich aber als teilweise problematisch. Neuere Studien entdeckten die Bedeutung von Fehlervermeidung und overfitting ebenso wie die Notwendigkeit der Validation.
Das generelle Schema, mutmaßliche Biomarker zu finden, ist folgendes: Zunächst müssen die Massenspektren vorbearbeitet werden. Dann können sie kodiert werden und eine alternative Darstellung der Daten liefern. Dazu wurde Diskrete Wavelet Transformation (DWT) verwendet. Als nächstes kann eine Auswahl oder Extraktion von Eigenschaften getroffen. Die Daten werden mit dem „Supervised Learning“-Ansatz klassifiziert. Eine hinreichend gute Klassifizierung erlaubt die Berücksichtigung von Peaks in dem Massenspektrum. Unter Verwendung von weiteren Messungen mit höherer Auflösung können die Peptide mit den Peaks in Verbindung gebracht werden.
In diesem Projekt kombinieren wir diskrete Wavelet-Transformation, statistische Verfahren für Feature-Auswahl und Support Vector Machines. Besondere Berücksichtigung findet die Generalisierbarkeit der produzierten Resultate, d.h. Robustness gegenüber Variabilität, die durch die Messzeitpunkte, das verwendete Laborinstrument oder Patienteneigenschaften (Alter, …). Die spezifischen Eigenschaften der berechneten Klassifikatoren werden mittels support Vectors und anderen Verfahren untersucht.

