BMBF-MALDI AMK: 3D-MALDI-Imaging zur Analyse proteomischer Marker und klinischer Wirkstofffverteilung
| Arbeitsgruppe: | AG Technomathematik |
| Leitung: | Prof. Dr. Dr. h.c. Peter Maaß ((0421) 218-63801, E-Mail: pmaass@math.uni-bremen.de ) |
| Bearbeitung: |
Dr. Fedor Alexandrov
Dr. Janina Oetjen Prof. Dr. Herbert Thiele Dr. Dennis Trede |
| Projektförderung: | BMBF |
| Projektpartner: | |
| Laufzeit: | 01.04.2011 - 31.07.2014 |
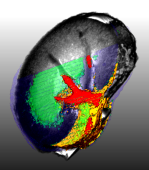
Überlagerung von 3D-MALDI-Imaging-Daten mit MRT-Daten
und mit mikroskopischen Bilddaten.
In sehr enger Kooperation mit medizinischen Partnern des Helmholtz Zentrum München und des Klinikums der Universität Jena werden klinisch bzw. pharmakologisch relevante Fragestellungen bearbeitet. Somit werden erstmals wichtige klinisch-onkologische Fragestellungen direkt in Organen und Geweben erforscht, die den Kontext des hochkomplexen, heterogenen 3D-Gewebeverbands voraussetzen. Zu diesen angegangenen Fragestellungen zählen beispielsweise die Verteilung von Tumorstammzellen, von Kopf-Halstumoren sowie die räumliche Wirkstoffverteilung und das Therapieansprechen beim Pankreaskarzinom.
Neben der Weiterentwicklung der mathematischen Algorithmen zur Vorverarbeitung, Daten-Analyse und statistischen Auswertung werden in Zusammenarbeit mit den Industriepartnern Bruker Daltonik GmbH und Human Solutions GmbH ebenfalls neue Konzepte für die Benutzerinteraktion entwickelt. Eine besondere Herausforderung ist dabei die 3D-Visualisierung und die direkte Interaktion mit diesen 3D-Daten. Da es sich bei den 3D-MALDI-Imaging-Aufnahmen um Daten handelt, die in jedem Raumpunkt ein 1D-Spektrum abbilden, sind hier klassische Visualisierungstechniken aus der medizinischen Bildverarbeitung nicht geeignet.
Eine reine Darstellung der 3D-Stoffwechsel-Information basierend auf den MALDI-Daten ist zwar als technisches Problem von hoher Komplexität, es ist jedoch aus diagnostischer Sicht für sich genommen noch wenig aussagekräftig. Zusammen mit dem Fraunhofer-Institut für Bildgestützte Medizin MEVIS werden daher diese Informationen zum Stoffwechsel mit anatomischer Information verknüpft, indem die MALDI-Daten mit Daten der Magnetresonanztomographie und mit mikroskopischen Bilddaten nach einer histologischen Färbung überlagert werden. Erst diese Verknüpfung erlaubt eine klinisch und pharmakologisch sinnvolle Auswertung für die Fragestellungen in der Onkologie.


