Wavelet-Cluster-Verfahren für die Analyse von Proteinspektren
| Arbeitsgruppe: | AG Technomathematik |
| Leitung: | Prof. Dr. Dr. h.c. Peter Maaß ((0421) 218-63801, E-Mail: pmaass@math.uni-bremen.de ) |
| Bearbeitung: | Prof. Dr. Kristian Bredies |
| Projektförderung: | Bremer Investitionssonderprogramm |
| Projektpartner: | Prof. Dr. Herbert Thiele, Bruker Daltonik GmbH, Bremen |
| Laufzeit: | seit 01.05.2003 |
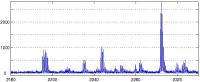
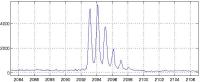
Die Überlebensrate bei den meisten Krebserkrankungen hängt entscheidend von einer zuverlässigen Früherkennung der Krankheit ab. Im Gegensatz zu krankheitsbedingten pathologischen Veränderungen, die über tomographische Verfahren erst vergleichsweise spät erkannt werden können, lassen sich Veränderungen der Proteinstruktur bereits im Frühstadium nachweisen. Deshalb werden Proteinprofile, die über Massenspektrometer aufgenommen werden, zunehmend im klinischen Einsatz als ergänzender und zuverlässiger Zusatzindikator im Frühstadium dieser Krankheit genutzt. Allerdings sind die kritischen Muster in den Proteinspektren, die eine Unterscheidung zwischen gesund und krankhaft liefern, nicht nur in den deutlichen Peaks der Spektren sondern gleichermaßen in der Form der Peaks und den feinskaligen Anteilen der Spektren enthalten. Ziel dieser Industriekooperation ist deshalb die Entwicklung von Wavelet-Cluster- Verfahren zur automatischen Auswertung derartiger Massenspektrometerdaten. Klassische Cluster-Verfahren für massenspektrometrische Daten basieren auf einer Bestimmung von Position und Fläche der wesentlichen Peaks. Die Feinstruktur des Spektrums wird bisher nicht berücksichtigt. Gemeinsam mit der Bruker Daltonik GmbH, einem weltweit führenden Produzenten von Massenspektrometern, wurde in diesem Projekt zunächst eine mathematische Modellierung der signifikanten Strukturen möglicher Spektren erarbeitet. Darauf aufbauend wurden Wavelet-Indikatoren zur Detektion kritischer Proteinmuster in Verbindung mit unterschiedlichen Klassifikationsmethoden (ClinProtTools, SVM) getestet. Dabei wurden für spezielle Krebsindikationen deutliche Verbesserungen erzielt. Dies wird aktuell in mehreren Schritten erweitert:
- Aufbau einer Datenbank mit Proteinspektren zur Früherkennung von Ovarian-Krebs.
- Erfassung kritischer Proteinmuster. Hier sollen erstmals Multiskalen-Deskriptoren entwickelt werden, die neben der Höhe der wesentlichen Proteinspektrallinien auch die Form der Peaks und die Struktur der feinskaligen Anteile der Spektren über geeignete Wavelet-Zerlegungen erfassen.
- Entwicklung und Implementierung einer darauf aufbauenden Wavelet-Cluster-Methode und Evaluation dieser Methode anhand von Testdaten.
- Kombination der Wavelet-Cluster-Methode mit bekannten Indikatoren.
- Implementierung des Gesamtverfahrens bei Bruker Daltonik.