Team für Deep Learning & Digitale Pathologie (DigiPath)
Das digipath-Team beschäftigt sich mit verschiedenen Herausforderung im Bereich der digitalen Pathologie. Dazu gehören zum Beispiel die Klassifizierung von Tumor- und nicht-Tumorgewebe oder von verschiedenen Tumortypen basierend auf digitalen Mikroskopiebildern. Es werden dazu klassische machine learning- als auch moderne deep learning-Methoden genutzt.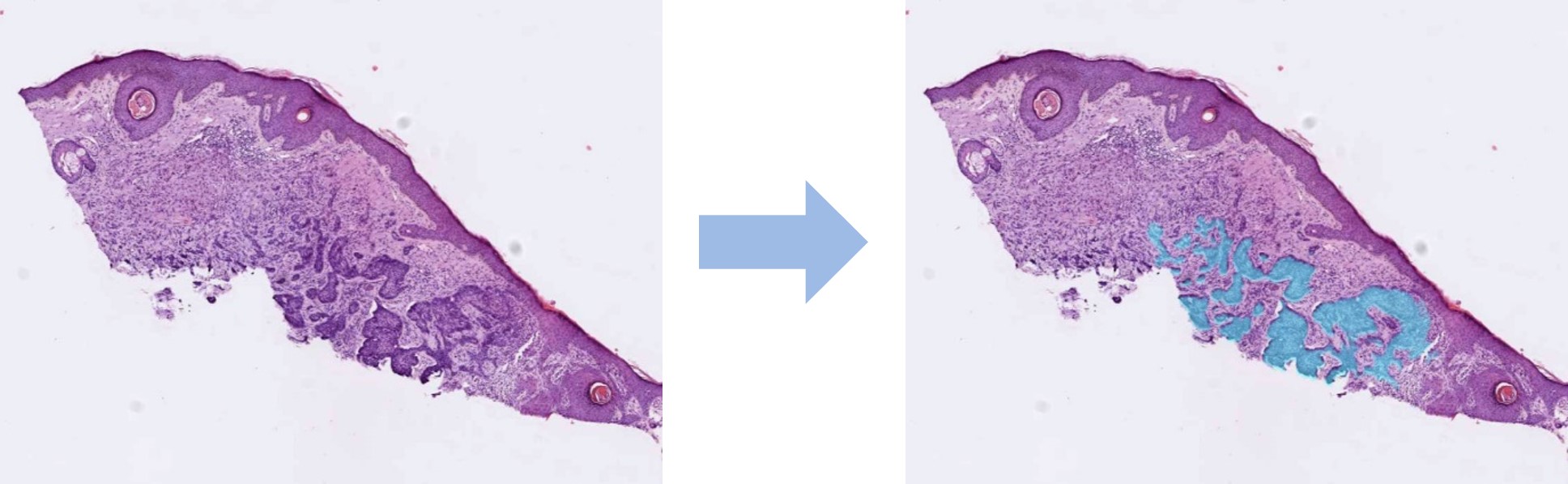
Projekte
- SPAplus - Small Data Probleme in der digitalen Pathologie und programmbegleitende Maßnahmen
- DIAMANT - Digitale Bildanalyse und bildgebende Massenspektrometrie zur Differenzierung von nichtkleinzelligem Lungenkrebs
- MALDISTAR - Studie zur Qualitätsbewertung, Standardisierung und Reproduzierbarkeit von Daten der bildgebenden MALDI-Massenspektrometrie
DigiPath - Viewer
Das digipath-Team entwickelt außerdem einen „DigiPath-Viewer“, eine Webanwendung, die zum Annotieren von digitalen Mikroskopiebildern und zum Anzeigen der Ergebnissen der Modelle genutzt werden kann. Der "DigiPath-Viewer" soll unsere Ergebnisse visualisieren und auch die Arbeit von Patholog*innen vereinfachen. Einige der Hauptfunktionen sind:- Integration mit Stylus-Stift für natürliche und präzise Annotationen.
- Ein halbautomatisches Annotationswerkzeug, das den Annotationsprozess beschleunigt. Das Annotationspolygon kann einfach durch Ziehen/Löschen der Punkte angepasst werden.
- Visualisierung der automatischen Tumorvorhersagen der künstlichen Intelligenz als Überlagerungen auf den Originalbildern. Die Transparenz des Overlays sowie die Schwelle zur Unterscheidung zwischen Tumor- oder Normalgewebe können leicht angepasst werden.
Teilnahme an Challenges
- CAMELYON17: Ziel dieser Challenge ist die Entwicklung einer Methode zur automatisierten Detektion und Klassifikation von Brustkrebsmetastasen in Whole-Slide bilder von histologischen Lymphknotenschnitten. Diese Aufgabe würde normalerweise eine umfangreiche mikroskopische Auswertung durch Pathologen erfordern. Daher wäre eine automatisierte Lösung sehr vielversprechend, um die Arbeitsbelastung der Pathologen zu verringern und gleichzeitig die Subjektivität bei der Diagnose zu reduzieren. Unser Team erreichte den 13. Platz im inoffiziellen Leaderboard, das auch nach den offiziellen Abgabeterminen offen blieb und mehr als 100 Einreichungen von den besten Unternehmen und Forschungsgruppen im Bereich der digitalen Pathologie erhalten hat.
Teamleiter
Das Team






Veröffentlichungen
- J. Behrmann, C. Etmann, T. Boskamp, R. Casadonte, J. Kriegsmann, P. Maaß.
Deep Learning for Tumor Classification in Imaging Mass Spectrometry.
Bioinformatics, 34(7):1215-1223, Oxford University Press, 2018. - C. Etmann, M. Schmidt, J. Behrmann, T. Boskamp, L. Hauberg-Lotte, A. Peter, R. Casadonte, J. Kriegsmann, P. Maaß.
Deep Relevance Regularization: Interpretable and Robust Tumor Typing of Imaging Mass Spectrometry Data.
Zur Veröffentlichung eingereicht.online unter: https://arxiv.org/abs/1912.05459