Bioinformatik-Optimierungssystem für Primer-Capture-Capture-Primer-Designs
| Arbeitsgruppe: | AG Technomathematik |
| Leitung: | Prof. Dr. Dr. h.c. Peter Maaß ((0421) 218-63801, E-Mail: pmaass@math.uni-bremen.de ) |
| Bearbeitung: | Dr. Manfred Nölte |
| Projektpartner: | SIRS-Lab GmbH, Jena |
| Laufzeit: | 01.07.2006 - 30.06.2008 |
Biochips werden zur Zeit mehr und mehr im Bereich der medizinischen Diagnostik eingesetzt. Mit deren Hilfe soll die Aktivität von Genen innerhalb kürzester Zeit erkannt und zur Frühdiagnostik und Therapiesteuerung genutzt werden.
Ausgangssituation. Microarrays (auch DNA-Chips genannt) erlauben eine nie zuvor möglich gewesene Geschwindigkeit in der Erstellung und Analyse biologischer Daten, da die Präsenz von Tausenden von Genen auf einem Microträger ein massiv paralleles Arbeiten ermöglicht.
Ausgangssituation. Microarrays (auch DNA-Chips genannt) erlauben eine nie zuvor möglich gewesene Geschwindigkeit in der Erstellung und Analyse biologischer Daten, da die Präsenz von Tausenden von Genen auf einem Microträger ein massiv paralleles Arbeiten ermöglicht.
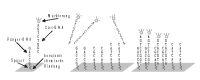
Auf einem Träger-Substrat werden strukturiert kurze DNA-Fragmente immobilisiert. Die auf der Matrix an einen bestimmten, d.h. bekannten Ort immobilisierte DNA bezeichnet man als Capture-DNA. Die zu untersuchende floureszent markierte Nukleinsäure wird als Target-DNA bezeichnet. Die Target DNA wird uber den Ort der Hybridisierung auf dem Chip identifiziert und anhand der Fluoreszenzstärke ggf. quantifiziert. Die Orte und die Intensität der Target-Hybridisierungen werden nach dem Abwaschen unspezifisch gebun- dener Targets uber Fluoreszenz-Scanning analysiert.
Vorarbeiten. Die SIRS-Lab GmbH, ein Biotechnologieunternehmen mit Sitz in Jena/Thüringen, erforscht u.a. mit Hilfe der DNA-Microarray-Technik den molekularen Hintergrund von akuten Entzündungsvorgängen. Dabei verfolgen sie das Ziel, neue Biomarker für die Früherkennung und Verlaufsbeobachtung von Infektionen und Sepsis zu entwickeln und diese für die spätere klinische Routinediagnostik nutzbar zu machen. Zwischen der SIRS-Lab GmbH und einer Ausgründung der Universität Bremen gab es bereits eine Zusammenarbeit im Bereich des Designs von Capture-Bibliotheken. An der Universität Bremen wurden im Rahmen des Centrums für angewandte Gensensorik (CAG) eine Dissertation „Optimierung von Oligonukleotid-Bibliothek für DNA-Mikroarrays“ und zwei Diplomarbeiten auf diesem Gebiet angefertigt. Weiterhin besteht eine Kooperation mit dem ttz/BIBIS (Bremerhavener Institut für Biologische Informationssysteme), welche zur Zeit im Rahmen des EU-Projektes „Nanoparts“ Capture-Sonden berechnen.
Projektziel. Die Optimierung von Capture-Bibliotheken, d.h. Sets von Captures, die hochspezifisch und -sensitiv sind, ist ein kombinatorisches Optimierungsproblem. Darüber hinaus wird das Optimierungsproblem noch komplexer, wenn neben der Auswahl geeigneter Capture-Nukleinsäuren Primer für die Targetvorbereitung mittels der Methode PCR(Polymerasekettenreaktion) ausgesucht werden. Bei der Auswahl der Primer und Captures sind Abhüngigkeiten zu berücksichtigen, wie z.B. die Lage auf der Target-Sequenz und der Abstand der Primer bzw. Capture sowie die Schnittmengen der falsch-positiven Sequenzübereinstimmungen. Projektziel ist die Entwicklung eines Systems zum optimierten Design von Primer-Capture-Capture-Primer-Experimenten. Zu einer gegebenen Spezifikation (Liste der Accession Numbers, Schmelztemperatur und Länge der Primer und der Capture, Amplifikat-Länge, Position der Captures, usw.) wird für jede Accession Number ein optimiertes Set von Primer und Captures berechnet.
Lösungsansatz.Aufbauend auf den oben genannten Vorarbeiten wird ein System für eine parallele Rechnerarchitektur auf der Basis von Linux/Unix entwickelt. Systemkomponenten sind:
Vorarbeiten. Die SIRS-Lab GmbH, ein Biotechnologieunternehmen mit Sitz in Jena/Thüringen, erforscht u.a. mit Hilfe der DNA-Microarray-Technik den molekularen Hintergrund von akuten Entzündungsvorgängen. Dabei verfolgen sie das Ziel, neue Biomarker für die Früherkennung und Verlaufsbeobachtung von Infektionen und Sepsis zu entwickeln und diese für die spätere klinische Routinediagnostik nutzbar zu machen. Zwischen der SIRS-Lab GmbH und einer Ausgründung der Universität Bremen gab es bereits eine Zusammenarbeit im Bereich des Designs von Capture-Bibliotheken. An der Universität Bremen wurden im Rahmen des Centrums für angewandte Gensensorik (CAG) eine Dissertation „Optimierung von Oligonukleotid-Bibliothek für DNA-Mikroarrays“ und zwei Diplomarbeiten auf diesem Gebiet angefertigt. Weiterhin besteht eine Kooperation mit dem ttz/BIBIS (Bremerhavener Institut für Biologische Informationssysteme), welche zur Zeit im Rahmen des EU-Projektes „Nanoparts“ Capture-Sonden berechnen.
Projektziel. Die Optimierung von Capture-Bibliotheken, d.h. Sets von Captures, die hochspezifisch und -sensitiv sind, ist ein kombinatorisches Optimierungsproblem. Darüber hinaus wird das Optimierungsproblem noch komplexer, wenn neben der Auswahl geeigneter Capture-Nukleinsäuren Primer für die Targetvorbereitung mittels der Methode PCR(Polymerasekettenreaktion) ausgesucht werden. Bei der Auswahl der Primer und Captures sind Abhüngigkeiten zu berücksichtigen, wie z.B. die Lage auf der Target-Sequenz und der Abstand der Primer bzw. Capture sowie die Schnittmengen der falsch-positiven Sequenzübereinstimmungen. Projektziel ist die Entwicklung eines Systems zum optimierten Design von Primer-Capture-Capture-Primer-Experimenten. Zu einer gegebenen Spezifikation (Liste der Accession Numbers, Schmelztemperatur und Länge der Primer und der Capture, Amplifikat-Länge, Position der Captures, usw.) wird für jede Accession Number ein optimiertes Set von Primer und Captures berechnet.
Lösungsansatz.Aufbauend auf den oben genannten Vorarbeiten wird ein System für eine parallele Rechnerarchitektur auf der Basis von Linux/Unix entwickelt. Systemkomponenten sind:
- BLAST-Datenbank
- Tools für die Berechnung der Oligonukleotid-Eigenschaften (Primer, Capture)
- kombinatorische Optimierung
- Dokumentation des Berechnungsergebnisses